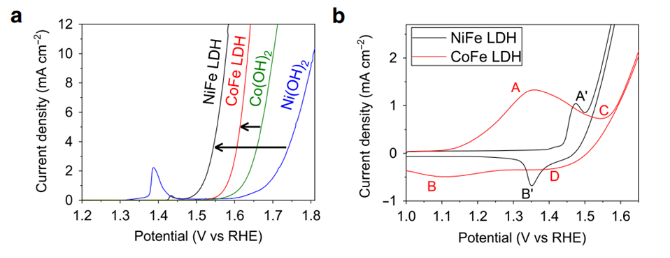
分享一篇發表在Nat Commun上的文章,文章題目為“Screening macrocyclic peptide libraries by yeast display allows control of selection process and affinity ranking”,文章通訊作者是威尼斯大學的Alessandro Angelini助理教授。

大環肽作為一種新興的藥物種類,具有較高的親和力和選擇性,且蛋白水解穩定性較好。目前已經開發了許多技術來發現新的大環肽配體,比如噬菌體展示、mRNA展示等。盡管這些方法能夠生成和篩選大型且結構多樣的大環肽庫,但是它們的篩選過程較為繁瑣,篩選過程中難以監測,得到候選分子的序列之后,需要經過后續的化學合成和純化步驟才能得到可用于親和力表征的肽配體。

針對這些缺點,作者開發了一個基于酵母展示的新型篩選策略,可以對篩選的過程進行調控,并對候選克隆進行親和力排序。該方法的核心是:先構建編碼結構多樣化的二硫鍵環化肽(包括單環、雙環的拓撲結構)的酵母展示文庫,在篩選的時候,先進行兩輪傳統的“結合-洗滌-洗脫-擴增”篩選過程,然后利用熒光激活細胞分選技術(FACS)對得到的酵母細胞進一步篩選,為了實現對篩選過程的調控,作者設計了一個雙色熒光標記系統,一個熒光探針用于檢測大環肽的表達水平(這里是檢測HA-tag來衡量大環肽的表達),另一個熒光探針用來檢測靶蛋白(靶蛋白被生物素修飾,故使用生物素的抗體來實現對其檢測)。通過這種雙色標記,FACS可以對酵母細胞進行逐一分析,并繪制“表達水平-結合能力”散點圖,從而篩選出那些與靶標結合能力較強的酵母細胞。篩選得到這些酵母細胞之后,作者一方面對它們的單克隆進行測序,另一方面將酵母克隆與一系列梯度稀釋的靶蛋白進行孵育,通過FACS檢測不同靶蛋白濃度下的結合熒光信號,可以繪制出每個克隆的結合等溫線,從而計算出表觀解離常數。
通過該方法,作者從文庫中針對5種完全不同的蛋白靶點,篩選得到了nM級甚至pM級的特異性大環肽配體,通過對其中一個最優分子與靶蛋白復合物的X射線晶體結構解析,作者從原子層面揭示了其高親和力的分子基礎,進一步驗證了該篩選方法的精準性和可靠性。
總而言之,本文將酵母展示技術與FACS結合,提供了一種快速發現大環肽配體的簡單、過程可視、結果可量化的新方案。
本文作者:LZ
責任編輯:WYQ
DOI:10.1038/s41467-025-60907-x
原文鏈接:https://doi.org/10.1038/s41467-025-60907-x