分享一篇發表在PNAS上的文章,文章題目為“Proximity-activated DNA scanning encoded sequencing for massive access to membrane proteins nanoscale organization”,本文通訊作者為西安交通大學的趙永席教授和趙越副教授、吉林大學化學院的孫穎教授。前兩位老師課題組的主要研究方向是高通量單分子與單細胞分析、核酸化學與生物學、功能納米材料與分子探針和微流控芯片;孫老師課題組的主要研究方向是生物化學傳感分析、傳感器界面組裝與修飾、微生物檢測。

膜蛋白在動態納米環境中表現出顯著的多樣性和精確的定位。傳統的方法受限于有限的蛋白質種類和空間覆蓋范圍,阻礙了對膜蛋白的全面分析,本文作者開發了一種名為proximity-a ctivated DNA scanning encoded sequencing(PADSE-seq)的方法,可以用于膜蛋白分布的大規模分析。
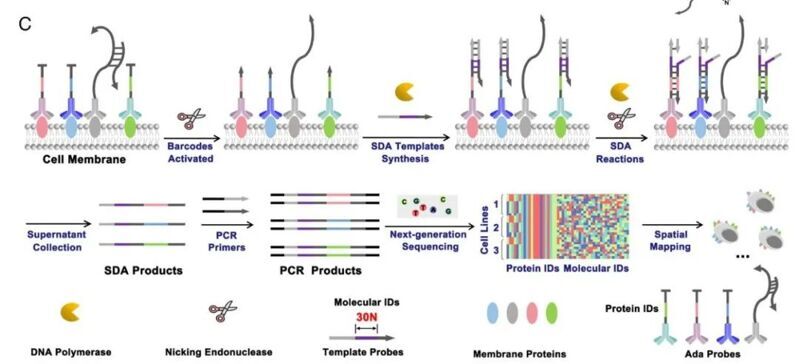
該方法的原理如下:固定在靶位點(T0)上的DNA掃描探針(SP)初始處于封閉狀態,需由切刻內切酶激活。激活后,SP能識別并依次激活其可接觸范圍內的相鄰位點(P1、P2、P3……Pn)上的DNA條形碼探針(BP)。由于切刻內切酶僅切割一條DNA鏈,SP可經歷多輪“雜交—切割—釋放”循環,從而逐步激活所有鄰近的BP。被激活的BP與帶有不同分子ID的模板探針(TP)結合,觸發聚合酶延伸并生成切刻內切酶識別位點,繼而觸發SDA反應,釋放出同時含有蛋白ID和分子ID的編碼產物。最終通過PCR和NGS對這些產物進行分析,從而獲取膜蛋白的空間分布信息。
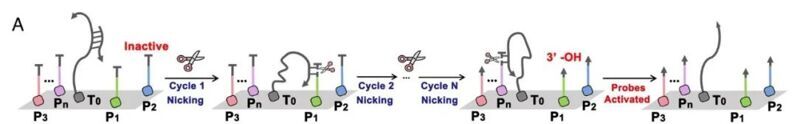
為了驗證該設想的可行性,作者先證明了使用切刻內切酶在溶液中連續激活BP的可行性,他們設計合成了用不同熒光團標記的探針SPsol、BPsol-Cy3和TPsol-Cy5,用PAGE分析每一步反應的產物,實驗結果表明每一步的產物均符合預期。之后作者使用序列長度或者序列作為條形碼來區分不同類型的BP,證明了PADSE-seq可以識別和激活BP并產生相應擴增產物。

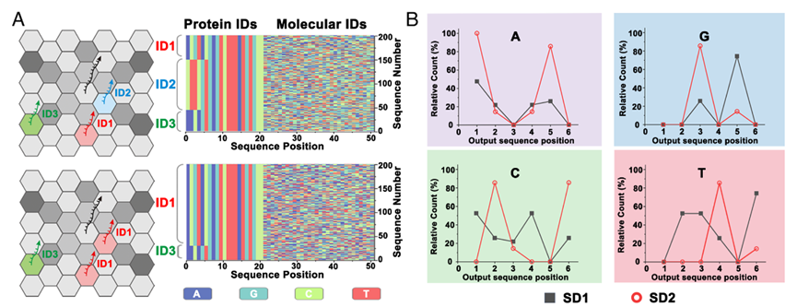
作者設計了兩種DNA折紙結構(SD1和SD2),用于模擬細胞膜蛋白在納米尺度上的空間分布差異。每個BPori探針包含兩個部分:用于標識蛋白種類的條形碼序列(蛋白質ID)以及一個30堿基的隨機序列(分子ID),后者用于追蹤和統計測序中每個分子的來源。在中心位點固定的SPori激活下,BPori探針通過SDA反應將蛋白質ID與分子ID連接,并經PCR擴增后進行高通量測序。對比分析發現:在SD1中,三種蛋白質ID對應的條形碼在測序結果中均有顯著出現;在SD2中,原位于第一圈的探針被另一種具有相同蛋白ID的探針替代后,ID2的條形碼顯著減少,而ID1的條形碼比例上升。進一步對前6個堿基(即蛋白質ID)進行統計分析,顯示出與探針空間分布相一致的條形碼比例變化。結果表明,測序信號與探針的空間位置高度相關,驗證了該方法在解析復雜膜蛋白鄰近關系方面的可行性。
之后,作為概念驗證,作者使用PADSE-seq繪制了乳腺癌細胞系中HER2附近實際中蛋白質的分布,證明了它在納米尺度上分析膜蛋白分布的能力。

總而言之,本文作者開發了一種名為PADSE-seq的技術,可以用于分析膜蛋白在空間中的分布。
本文作者:LZ
責任編輯:MB
原文鏈接:https://doi.org/10.1073/pnas.2425000122